Aula 16 – Tensor Flow – Redes Neurais – Classificação com Estimator API
Aula 16 – Tensor Flow – Redes Neurais – Classificação com Estimator API
Voltar para página principal do blog
Todas as aulas desse curso
Aula 15 Aula 17
Meu github:
https://github.com/toticavalcanti
Documentação oficial do TensorFlow:
https://www.tensorflow.org/
Link para o notebook da aula:
notebook-da-aula
Quer aprender python3 de graça e com certificado? Acesse então:
https://workover.com.br/python-codigo-fluente

Python com Tensorflow
Se gostarem do conteúdo dêem um joinha  na página do Código Fluente no
na página do Código Fluente no
Facebook
https://digitalinnovation.one/
Link do código fluente no Pinterest
Aproveito para deixar meus link de afiliados:
Hostinger
Digital Ocean
One.com
Toti:
https://www.youtube.com/channel/UCUEtjLuDpcOvR3mIUr-viOA
Backing track / Play-along:
https://www.youtube.com/channel/UCT3TryVMqTqYBjf5g5WAHfA
Código Fluente
https://www.youtube.com/channel/UCgn-O-88XBAwdG9gUWkkb0w
Putz!
https://www.youtube.com/channel/UCZXop2-CECwyFYmHbhnAkAw
Fiquem a vontade para me adicionar ao linkedin.
Aula 16 – Tensor Flow – Redes Neurais – Classificação com Estimator API
Nessa aula iremos construir um modelo classificador de rede neural profunda ou densa ( DNN ) usando o DNNClassifier do tensorflow.
Lembrando que uma rede neural profunda (DNN) é uma rede neural artificial (ANN) com várias camadas entre as camadas de entrada e saída.
A base de dados que iremos usar é o: Pima Indians Diabetes
É uma classificação binária, 1 se o paciente tem diabetes e 0 se o paciente não tem diabetes.
Contexto
Esse conjunto de dados é originalmente do Instituto Nacional de Diabetes e Doenças Digestivas e Renais.
O objetivo do conjunto de dados é prever diagnosticamente se um paciente tem diabetes ou não, com base em certas medidas diagnósticas incluídas no conjunto de dados.
Várias restrições foram colocadas na seleção dessas instâncias de um banco de dados maior.
Em particular, todos os pacientes aqui são mulheres com pelo menos 21 anos de herança indígena Pima.
Os Pima são índios norte-americanos que tradicionalmente viviam ao longo dos rios Gila e Salt no Arizona, EUA.
Conteúdo
O conjunto de dados consiste em várias variáveis preditoras médicas e uma variável de destino, ou resultado.
As variáveis preditoras incluem o número de gestações que a paciente teve, seu IMC, nível de insulina, idade e assim por diante.
Variáveis ou Features
Pregnancies: Número de gestações
Glucose: concentração de glicose no plasma ao longo de 2 horas em um teste de tolerância à glicose oral
BloodPressure: pressão arterial diastólica (mm Hg)
SkinThickness: espessura da dobra da pele do tríceps (mm)
Insulin: insulina sérica de 2 horas (mu U / ml)
BMI: IMC, índice de massa corporal (peso em kg / (altura em m) 2)
DiabetesPedigreeFunction: função de linhagem de diabetes (uma função que avalia a probabilidade de diabetes com base no histórico familiar)
Age: Idade (anos)
Outcome: variável de classe (0 se não diabético, 1 se diabético)
Normalização
O objetivo da normalização é alterar os valores das colunas numéricas no conjunto de dados usando uma escala comum, sem distorcer diferenças nos intervalos de valores ou perda de informações evitando que o modelo fique enviesado.
Imagine por exemplo que você tenha uma coluna variando entre 0 e 1, uma outra entre -10 e 10, uma outra entre 1000 e 10000, essa grande diferença na escala pode causar problemas quando você tenta combinar os valores.
Logo aqui abaixo, tem o código do notebook da aula, todo comentado.
# -*- coding: utf-8 -*-
"""04-neural-network-classification.ipynb
Automatically generated by Colaboratory.
Original file is located at
https://colab.research.google.com/drive/1xeBEsdArLqHNB4cRQdUZsZisLrkrfZfK
# Tensorflow 1
## Aula 16 - Tensor Flow - Redes Neurais - Classificação com DNNClassifier
Trabalharemos com uma base de dados real, é o Pima Indians Diabetes Database
Usaremos o classificador de rede neural densa ( DNNClassifier )
### Importa o tensorflow
"""
import tensorflow.compat.v1 as tf
tf.disable_v2_behavior()
"""## Importa o numpy, o pandas e o matplotlib"""
# Commented out IPython magic to ensure Python compatibility.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
# %matplotlib inline
"""## Leitura dos dados e análise exploratória"""
# Nome para a coluna ou mapa de recursos
columns_to_named = ["Pregnancies","Glucose","BloodPressure",
"SkinThickness","Insulin","BMI","DiabetesPedigreeFunction",
"Age","Class"]
# Ler o conjunto de dados e renomeia a coluna
df = pd.read_csv(
"https://raw.githubusercontent.com/toticavalcanti/pima-indians-diabetes/master/diabetes.csv",
header = 0,
names = columns_to_named)
# Resumo dos dados
df.describe()
# Contagem de valores nulos
df.isnull().sum()
corr = df.corr()
print(corr)
sns.heatmap(corr,
xticklabels=corr.columns,
yticklabels=corr.columns)
"""No mapa de calor acima, as cores mais brilhantes indicam mais correlação.
Como podemos ver na tabela e no mapa de calor, os níveis de glicose, idade, IMC e número de gestações têm correlação significativa com a variável de resultado, Outcome, que mudei o nome para Class.
Observe também a correlação entre pares de características, como idade e gravidez, ou insulina e espessura da pele.
## Diabéticos e não diabéticos
"""
# Gráfico de contagem de resultados
sns.countplot(x = 'Class',data = df)
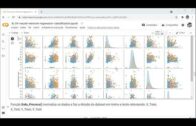
"""### Histograma de cada característica"""
# Histograma de cada característica
import itertools
col = df.columns[:8]
plt.subplots(figsize = (20, 15))
length = len(col)
for i, j in itertools.zip_longest(col, range(length)):
plt.subplot((length/2), 3, j + 1)
plt.subplots_adjust(wspace = 0.1,hspace = 0.5)
df[i].hist(bins = 20)
plt.title(i)
plt.show()
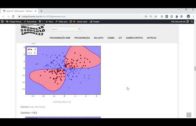
"""### Gráfico de pares"""
sns.pairplot(data = df, hue = 'Class')
plt.show()
"""### Função Data_Process() normaliza os dados e faz a divisão do dataset em treino e teste retornando: X_Train, X_Test, Y_Train, Y_Test
## Importa o train_test_split
"""
from sklearn.model_selection import train_test_split
def Data_Process():
col_norm =['Pregnancies', 'Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI', 'DiabetesPedigreeFunction']
# Normalização usando uma função Lambda personalizada
df1_norm = df[col_norm].apply(lambda x :( (x - x.min()) / (x.max()-x.min()) ) )
X_Data = df1_norm
Y_Data = df["Class"]
X_Train, X_Test, Y_Train, Y_Test = train_test_split(X_Data,Y_Data, test_size=0.3,random_state=101)
return X_Train, X_Test, Y_Train, Y_Test
"""## Função create_feature_column() retorna as colunas de recursos, ou seja, as features"""
def create_feature_column():
feat_Pregnancies = tf.feature_column.numeric_column('Pregnancies')
feat_Glucose = tf.feature_column.numeric_column('Glucose')
feat_BloodPressure = tf.feature_column.numeric_column('BloodPressure')
feat_SkinThickness_tricep = tf.feature_column.numeric_column('SkinThickness')
feat_Insulin = tf.feature_column.numeric_column('Insulin')
feat_BMI = tf.feature_column.numeric_column('BMI')
feat_DiabetesPedigreeFunction = tf.feature_column.numeric_column('DiabetesPedigreeFunction')
feature_column = [feat_Pregnancies, feat_Glucose, feat_BloodPressure,
feat_SkinThickness_tricep, feat_Insulin,
feat_BMI , feat_DiabetesPedigreeFunction]
return feature_column
"""## Divide os dados em 80% dos dados para treino e 30% para teste através da função Data_Process() """
X_Train, X_Test, Y_Train, Y_Test = Data_Process()
"""## Chama a função que definimos mais acima create_feature_column() que retorna as features"""
feature_column = create_feature_column()
"""## O pandas_input_fn() retorna a função de entrada que vai alimentar o DataFrame Pandas no modelo
Vamos usar a input_func para treinar o modelo e a eval_func para testar o modelo
"""
input_func = tf.estimator.inputs.pandas_input_fn(X_Train,
Y_Train,
batch_size = 50,
num_epochs = 1000,
shuffle = True)
eval_func = tf.estimator.inputs.pandas_input_fn(X_Test,
Y_Test,
batch_size = 50,
num_epochs = 1,
shuffle = False)
predict_input_fn = tf.estimator.inputs.pandas_input_fn(
x = X_Test,
num_epochs = 1,
shuffle = False)
"""## Cria o modelo DNN ( Deep Neural Network )"""
dnnmodel = tf.estimator.DNNClassifier(
hidden_units = [20,20],
feature_columns = feature_column,
n_classes = 2,
activation_fn = tf.nn.softmax,
dropout = None,
optimizer = tf.train.AdamOptimizer(learning_rate=0.01)
)
"""## Treino do modelo"""
history = dnnmodel.train(input_fn = input_func, steps = 500)
"""## Teste do modelo"""
dnnmodel.evaluate(eval_func)
"""## Predições"""
from sklearn.metrics import confusion_matrix, classification_report
predictions = list(dnnmodel.predict(input_fn = predict_input_fn))
prediction = [p["class_ids"][0] for p in predictions]
data = classification_report(Y_Test, prediction)
conmat = confusion_matrix(Y_Test, prediction)
"""## Análise
## Instala no notebook o scikit-plot
OBS. Basta rodar uma vez, por isso tá comentado, se quiser instalar é só descomentar
"""
!pip install scikit-plot
import scikitplot as skplt
skplt.metrics.plot_confusion_matrix(Y_Test,
prediction,
figsize = (6,6),
title = "Confusion Matrix")
print(data)Função de custo
Antes de seguir para as métricas, vamos lembrar que a função de custo, ou de perda ( loss ), é o que queremos minimizar treinando a rede, afim de que ela faça seu trabalho corretamente, que no nosso caso é dizer se um paciente tem diabetes ou não baseado nas features que os dados oferece.
Métricas
accuracy = (TP + TN)/(TP + TN + FP + FN)
precision = TP / (TP + FP)
recall = TP / (TP + FN)
f1-score = 2 * precision * recall / precision + recall = TP / TP + 0.5 * (FP + FN)
Onde: TP = True positive; FP = False positive; TN = True negative; FN = False negative
O f1-score na análise estatística da classificação binária, é uma medida da precisão de um teste. É calculado a partir da precisão e recuperação do teste
A precision é definida como ‘a qualidade de ser exato‘ e refere-se a quão próximas duas ou mais medições estão umas das outras, independentemente de essas medições serem precisas ou não. É possível que as medições de precisão não sejam precisas.
O support é número de ocorrências de cada rótulo em y_true. No caso do diabetes dataset, a quantidade de pessoas com diabetes e sem diabetes.
A precision / mean: média da precisão
A accuracy é a porcentagem do número correto de classificações. Lembrando que é necessário que a proporção de exemplos para cada classe sejam iguais, é uma boa métrica para quando as penalidades de acerto e erro para cada classe forem as mesmas.
A accuracy é definida como ‘o grau em que o resultado de uma medição está em conformidade com o valor correto ou padrão‘ e, essencialmente, refere-se a quão próxima uma medição está de seu valor acordado.
accuracy_baseline: linha base da precisão com base na média dos rótulos. Isso é o melhor que o modelo pode fazer ao prever uma classe sempre.
auc ou área sob a curva (ROC): informa algo sobre a taxa de verdadeiro / falso positivo. Resumindo, é uma métrica interessante para tarefas com classes desproporcionais.
auc_precision_recall: É a porcentagem de instâncias relevantes, entre as instâncias recuperadas, que foram recuperadas sobre a quantidade total de instâncias relevantes.
Quando você treina uma rede, geralmente a alimenta com insumos em lotes.
No exemplo, o tamanho do lote é 50, então a loss é a soma das perdas em um lote, enquanto a perda média é a média das perdas em todos os lotes.
Average_loss: Você geralmente está minimizando alguma função de custo, e este é o valor médio de perda dessa função de custo em todos os lotes.
loss: O valor atual da perda do lote atual, ou seja, a soma da perda do último lote.
global_step: Número de iterações
label/mean: O label é o valor que a rede deve produzir, ou seja, prever para uma determinada entrada. Resumindo, é o label dividido pelo número de elementos.
prediction/mean: é a soma de todas essas saídas dividida pelo número de saídas.